Одной из наиболее важных особенностей языка R является его расширяемость. Много разных учёных создали пакеты для R и выложили их в открытом доступе (архив CRAN). Среди прочих там есть и пакет network, который можно установить, набрав в командной строке R (при подключённом интернете!)
>install.packages ('network') После того, как пакет установлен, его следует запустить командой
>library(network)Сеть данных
Рассмотрим небольшую часть огромной сети, состоящей из 10000 узлов, в котором рёбра означают (гетеро)сексуальные отношения между соответствующими узлами. Поскольку контакты женщина-женщина и мужчина-мужчина из этой сети убраны с целью её упрощения, рассматриваемую сеть можно считать бимодальной (т. е. такой, узлы в которой условно делятся на два подмножества, внутри которых нет рёбер). Все контакты (рёбра нашего графа) являются неориентированными. В начале этого моделирования 5 случайно выбранных «семян» (четверо белых мужчин и один чёрный мужчина) инфицированы какой-то гипотетической ЗППП, которая может со 100%-й вероятностью передаваться при контакте. В нашей модели есть только две расы, при этом пропорции полов и рас среди 10000 узлов сети были выбраны так, чтобы репрезентировать определённый набор данных. В конце моделирования, соответствовавшего 3600 дням, были инфицированы 256 узлов.
У каждого узла есть несколько связанных с ним атрибутов. Введём их в R помощью команды read.table:
>help(read.table) # Взгляните на варианты. Обратите внимание на значения по умолчанию.
>nodeinfo=read.table("http://www.stat.psu.edu/~dhunter/Rnetworks/nodal.attr.txt",head=T)Мы можем использовать объект nodeinfo, который мы только-что создали, чтобы узнать характеристики этих акторов — но об этом позже. Для начала давайте взглянем на список рёбер в нашей сети:
>myedges=read.table("http://www.stat.psu.edu/~dhunter/Rnetworks/edgelist.txt") Объект myedges представляет собой матрицу из двух столбцов. В каждой строке женщинам и мужчинам (узлам сети) присвоены идентификаторы узла. Мы можем узнать размеры этой матрицы:
>dim(myedges) Теперь давайте узнаем немного больше об объекте nodeinfo. Названия столбцов:
>names(nodeinfo) Мы можем, например, ещё определить число людей каждой расы в этой сети:
>table(nodeinfo$race) # Эти все 3 команды эквивалентны
>table(nodeinfo[,2])
>table(nodeinfo[,"race"]) Или же мы можем посмотреть двумерную таблицу раса-пол:
>table(sex$nodeinfo,race$nodeinfo) # Такую таблицу трудно читать, поэтому нужно добавить заголовки:
>table(sex=nodeinfo$sex, race=nodeinfo$race) В R есть потрясающие графические возможности (нужно, правда, потратить много времени на изучение всех тонкостей так, чтобы виртуозно манипулировать ими и получать на выходе именно то, что Вам нужно). Мы, напр., можем сделать гистограмму времени заражения или сравнить время заражения по признаку расы с помощью команды boxplots:
>boxplot(nodeinfo$time~nodeinfo$race)Создание сети объектов
Помните команду library(network)? Теперь у нас есть ряд инструментов, с помощью которых мы можем работать на сетевых объектах. Давайте начнём с создания такого объекта:
>diseasenw=network(myedges) Мы можем построить эту сеть:
>plot(diseasenw) 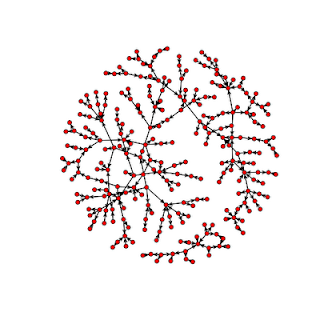
Заметьте, что наличие стрелок на рисунке указывает на то, что наша сеть является направленной (построение направленных сетей задано в функции network() по умолчанию, но это легко изменить). Кроме того, давайте включим в рисунок чуть больше информации об аттрибутах узлов и укажем, что наша сеть бимодальна. В дальнейшем, мы проведём разделительную линию между узлами одного типа и узлами другого типа, в этом случае такая граница пролегает по самому большому номеру узла женского пола (т. е. для того, чтобы указать программе, что сеть бимодальна, нужно указать тот номер, до которого узлы будут считаться женскими, а после которого — мужскими):
>diseasenw=network(myedges,directed=F,bipartite=132,vertex.attr=nodeinfo)Построим рисунок снова:
>plot(diseasenw)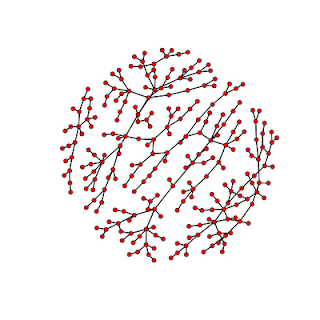
Можно ещё очень многое поменять на полученном рисунке. Для того, чтобы узнать об этих возможностях, следует познакомиться с документацией для команды plot (наберите help(plot) в командной строке R).
Например, если нам нужно цветом узлов обозначить расы, а их формой — пол, то следует записать:
>plot(diseasenw, vertex.col=3-nodeinfo$race,vertex.sides=2+nodeinfo$sex,main="Кружки суть женщины, треугольники — мужчины")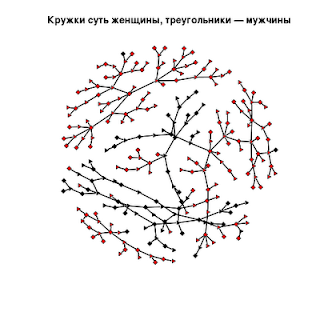
Немає коментарів:
Дописати коментар